- [ 김현욱 교수 연구실 ] 암 유발 물질 컴퓨터로 예측하다
- 관리자 |
- 2024-03-18 09:53:01|
- 709
- 2024-03-18 09:53:01|
- 관련보도링크
▶ 아래 내용은 우리 학교 홍보실에서 작성하여 학교 홈페이지에 게시한 자료를 인용하며, 헤럴드경제, 대학저널, 메디컬투데이 등 여러 매체에 소개되었습니다.

< (왼쪽부터) 생명화학공학과 김현욱 교수, 하버드 의과대학 이가령 박사, 하버드 의과대학 이상미 박사, 서울대학교병원 윤홍석 교수, 서울대학교병원 고영일 교수 >
암은 정상세포와 다르게 세포 내 비정상적인 축적을 통해 유발되는 대사 반응을 하며, 암의 치료 및 진단을 목적으로 이런 암 대사반응에 대해 다방면으로 연구되고 있다. 이에 우리 대학 연구진이 컴퓨터를
통해 24개 암종에 해당하는 1,043명의 암 환자에 대한 대사 모델 구축에 성공했다.
우리 대학 생명화학공학과 김현욱 교수, 이상엽 특훈교수 연구팀이 서울대학교병원 고영일 교수, 윤홍석 교수 및 정창욱 교수 연구팀과의 공동연구를 통해, 암 체세포 유전자 돌연변이와 연관된 새로운 대사
물질 및 대사경로를 예측하는 컴퓨터 방법론을 개발했다고 18일 밝혔다.
최근 암 유발 대사물질(oncometabolite)*의 발견과 이를 표적으로 하는 신약들이 미국식품의약국(FDA)의 승인을 받으며 주목받고 있는데, 이에는 급성 골수성 백혈병의 치료제로 사용되고 있는 ‘팁소보(성분명:
아이보시데닙)’ 및 약물 ‘아이드하이파(성분명: 에나시데닙)’가 포함된다.
* 암 유발 대사물질 (oncometabolite) : 세포 내 비정상적인 축적을 통해 암을 유발하는 대사물질. 이러한 대사물질들은 특정 유전자 돌연변이의 영향으로 대사 과정 중에 비정상적으로 높은 농도로 축적되며,
이러한 축적은 암세포의 성장과 생존을 촉진함. 기존 연구에서 확인된 주요 암 유발 대사물질로는 2-하이드록시글루타레이트(2-hydroxyglutarate), 숙시네이트(succinate), 푸마레이트(fumarate) 등이 보고됨.
하지만, 암 대사 연구와 새로운 암 유발 대사물질 발굴에는 대사체학 등의 방법론이 필요하며, 이를 대규모 환자 샘플에 적용하기 위해서는 상당한 시간과 비용이 소요된다. 이러한 이유로, 암과 관련된 많은
유전자 돌연변이들이 밝혀졌음에도, 그에 상응하는 암 유발 대사물질은 극소수만 알려져 있다.
김현욱 교수 공동연구팀은 세포 대사 정보를 예측할 수 있는 게놈 수준의 대사 모델*에 국제 암 연구 컨소시엄에서 공개하고 있는 암 환자들의 전사체 데이터를 통합해, 24개 암종에 해당하는 1,043명의 암
환자에 대한 대사 모델을 성공적으로 구축했다.
* 게놈 수준의 대사모델: 세포의 전체 대사 네트워크를 다루는 컴퓨터 모델로서, 세포 내 모든 대사반응에 대한 정보가 담겨 있으며, 다양한 조건에서 세포의 대사 활성을 예측하는 것이 가능
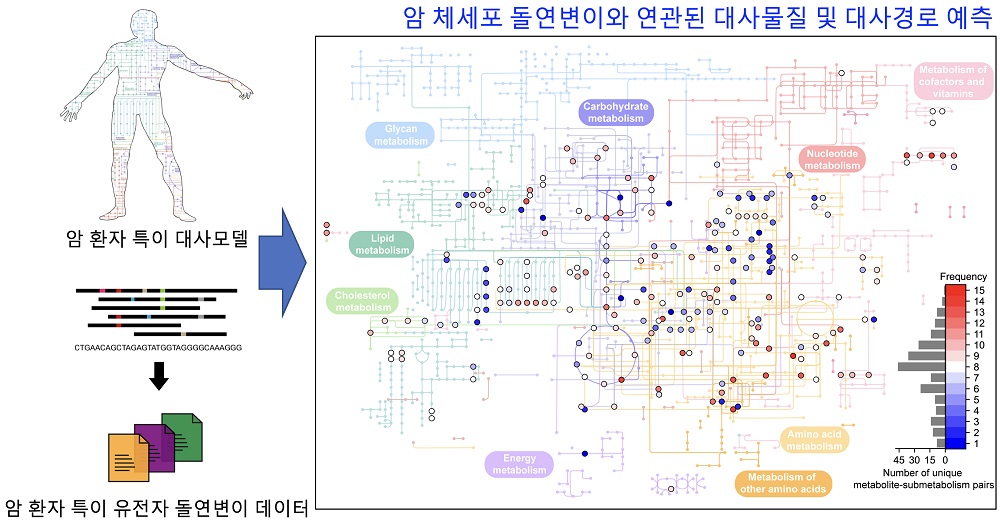
< 그림 1. 암 체세포 돌연변이와 연관된 대사물질 및 대사경로를 예측하는 컴퓨터 방법론 모식도 >
공동연구팀은 1,043명의 암 환자 특이 대사 모델과 동일 환자들의 암 체세포 돌연변이 데이터를 활용해, 다음의 4단계로 구성된 컴퓨터 방법론을 개발했다 (그림 1). 첫 단계에서는 암 환자 특이 대사 모델을
시뮬레이션해, 환자 별로 모든 대사물질들의 활성을 예측한다. 두 번째 단계로는 특정 유전자 돌연변이가 앞서 예측된 대사물질의 활성에 유의한 차이를 일으키는 짝을 선별한다. 세 번째 단계로, 특정 유전자
돌연변이와 연결된 대사물질들을 대상으로, 이들과 유의하게 연관된 대사경로를 추가로 선별한다. 마지막 단계로서, ‘유전자-대사물질-대사경로’ 조합을 완성해, 컴퓨터 방법론 결과로써 도출하게 된다.
이번 논문의 공동 제1 저자인 이가령 박사(現 다나파버 암센터 및 하버드 의과대학 박사후연구원)와 이상미 박사(現 하버드 의과대학 박사후연구원)는 “이번 연구에서 개발된 방법론은 암 환자 코호트의 돌연
변이 및 전사체 데이터를 토대로 다른 암종에 대해서도 쉽게 적용될 수 있으며, 유전자 돌연변이가 대사경로를 통해 어떻게 세포대사에 변화를 일으키는지 체계적으로 예측할 수 있는 최초의 컴퓨터 방법론
이라는 데 큰 의의가 있다” 한다고 말했다.
또한 김현욱 교수는 “이번 공동연구의 결과는 향후 암 대사 및 암 유발 대사물질 연구에서 중요한 참고 자료로 활용될 수 있을 것”이라고 강조했다.
한편 이번 논문은 바이오메드 센트럴(BioMed Central) 社가 발행하며, 생명공학 및 유전학 분야의 대표적 국제학술지인 게놈 바이올로지(Genome Biology, JCR 분야 상위 5% 이내)에 게재됐다.
※ 논문명 : Prediction of metabolites associated with somatic mutations in cancers by using genome-scale metabolic models and mutation data
※ 저자 정보 : 이가령(한국과학기술원, 공동 제1 저자), 이상미(한국과학기술원, 공동 제1 저자), 이성영(서울대학교병원, 공동저자), 정창욱(서울대학교병원, 공동저자), 송효진(서울대학교병원, 공동저자),
이상엽(한국과학기술원, 공동저자), 윤홍석(서울대학교병원, 교신저자), 고영일(서울대학교병원, 교신저자), 김현욱(한국과학기술원, 교신저자) 포함 총 9명
이번 연구는 과학기술정보통신부 한국연구재단의 지원을 받아 수행됐다.
| 첨부파일 |
|
|---|
- 이전
- [ 최남순 교수 연구실 ] 세계 최고 수준 리튬 금속배터리 용매 개발
- 2024-03-20
- 다음
- 이상엽 특훈교수 / (논단) 김치피자와 K푸드 발전 전략
- 2024-03-11

