- [ 이상엽 특훈교수 연구실 ] 숨겨진 효소 쏙쏙 찾아내는 인공지능 개발
- 관리자 |
- 2023-11-24 11:12:12|
- 377
- 2023-11-24 11:12:12|
- 관련보도링크
▶ 아래 내용은 우리 학교 홍보실에서 작성하여 학교 홈페이지에 게시한 자료이며, 관련기사는 헤럴드경제, 한국강사신문, 동아사이언스, 이데일리 등 여러 매체에 소개되었습니다.

< (왼쪽부터) 생명화학공학과 이상엽 특훈교수, 김기배 박사과정 >
대장균은 가장 많이 연구된 생명체 중 하나에 해당되지만 아직 대장균을 구성한 단백질 30%의 기능에 대해 명확하게 밝혀지지 않았다. 이에 대해 인공지능을 활용하여 아직 명확하게 밝혀진 바 없던 단백질에서
464종의 효소를 발견하였으며, 이 중 3종의 단백질의 예측된 기능을 시험관 내 효소 분석 방법을 통해 검증하는데 성공하였다.
우리 대학 생명화학공학과 이상엽 특훈교수와 캘리포니아대학교 샌디에이고(UCSD) 생명공학과 버나드 펄슨(Bernhard Palsson) 교수 공동연구팀이 단백질 서열을 활용, 해당 단백질의 효소 기능을 예측할 수
있는 인공지능, `딥 EC 트랜스포머(DeepECtransformer)'를 개발해 빠르고 정확하게 효소 기능을 파악할 수 있는 예측 시스템을 구축했다고 24일 밝혔다.
효소는 생물학적 반응을 촉매하는 단백질로서, 생명체 내 존재하는 다양한 화학 반응과 이에 따라 결정되는 생명체의 대사 특성을 파악하기 위해서는 각 효소의 기능을 이해하는 것이 필수적이다. EC 번호(효소
고유 번호, Enzyme Commission number)는 국제생화학 및 분자 생물학연맹 (International Union of Biochemistry and Molecular Biology, IUBMB)가 고안한 효소 기능 분류 체계로서, 다양한 유기체의 대사
특성을 이해하기 위해선 게놈 서열에서 존재하는 효소의 종류와 EC 번호를 빠르게 분석할 수 있는 기술 개발이 필요하다.
단백질의 기능 및 효소 기능 예측을 위해 인공지능을 활용하는 다양한 예측 시스템 또한 보고됐지만, 인공지능의 추론 과정을 직접 확인할 수 없는 블랙박스(black box)의 특징을 가졌거나, 효소 서열 내 아미
노산 잔기(최소 단위) 수준으로 해석하지 못하는 문제가 있었다. 공동연구팀은 심층학습 기법과 단백질 상동성 분석 모듈을 활용해 주어진 단백질 서열의 효소 기능을 예측하는 인공지능 딥 EC 트랜스
포머(DeepECtransformer)를 개발했다. 연구팀은 이번 연구에서 더 다양한 효소 기능을 정확하게 예측할 수 있도록 단백질 서열 전체 문맥에서 효소 기능에 중요한 정보를 추출하였고, 이를 통해 효소의 EC
번호를 정확하게 예측할 수 있었다. 개발된 인공지능은 총 5,360종류의 EC 번호를 예측할 수 있었다.
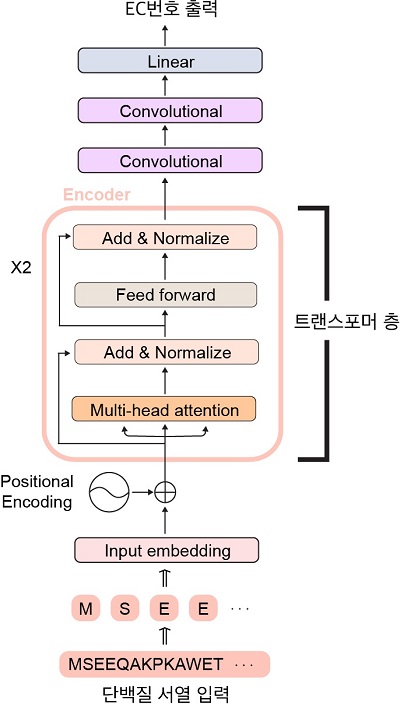
< 그림 1. DeepECtransformer의 인공 신경망 구조 >
공동연구팀은 나아가 딥 EC 트랜스포머의 인공신경망 내 정보 흐름을 분석하여 인공지능이 추론 과정에서 효소 기능에 중요한 활성 부위나 보조 인자 결합 부위 정보를 활용하고 있음을 밝혀냈다. 이처럼
인공지능의 블랙박스를 해석함으로써 인공지능이 학습 과정에서 스스로 효소 기능에 중요한 특징을 파악하고 있음을 연구팀은 확인했다.
이번 논문의 제1 저자인 우리 대학 김기배 박사과정생은 “이번에 개발한 예측 시스템을 활용해 아직 밝혀진 적 없던 효소의 기능을 새롭게 예측하고 실험으로 검증할 수 있었다”고 말했다. 그는 또한 “딥 EC 트랜스
포머를 활용해 생명체 내 밝혀지지 않았던 효소를 파악함으로써 유용 화합물을 생합성하기 위해 필요한 효소나 플라스틱을 생분해하기 위해 필요한 효소 등 다양한 대사 과정을 새롭게 밝혀낼 수 있을 것”이라고
덧붙였다.
또한 이상엽 특훈교수는 “효소 기능을 빠르고 정확하게 예측하는 딥 EC 트랜스포머는 기능 유전체학의 핵심 기술로서 시스템 수준에서 전체 효소들의 기능들을 분석할 수 있게 한다”며 “이를 활용해 모든 효소
정보를 포함한 대사 네트워크를 기반으로 친환경 미생물 공장 개발을 수행할 수 있을 것”이라고 밝혔다.
생명화학공학과 김기배 박사과정이 참여한 이번 논문은 국제 학술지 네이처(Nature) 誌가 발행하는 `네이처 커뮤니케이션즈(Nature Communications)'에 동료 심사를 거쳐 11월 14일 字 게재됐다.
※ 논문명 : 트랜스포머 레이어와 딥러닝을 사용하여 효소 인코딩 유전자의 기능적 주석 달기 (Functional annotation of enzyme-encoding genes using deep learning with transformer layers)
※ 저자 정보 : 김기배 (한국과학기술원, 제1 저자), 김지연 (한국과학기술원, 제2 저자), 이종언 (한국과학기술원, 제3 저자), Charles J. Norsigian (UCSD, 제4 저자), Bernhard O. Palsson (UCSD, 제5 저자) 및
이상엽(한국과학기술원, 교신저자) 포함 총 6 명
한편, 이번 연구는 과기정통부가 지원하는 ‘석유대체 친환경 화학기술개발사업의 ‘바이오화학산업 선도를 위한 차세대 바이오리파이너리 원천기술 개발’ 과제(과제책임자 KAIST 이상엽 특훈교수)의 지원을 받아
수행됐다.
| 첨부파일 |
|
|---|
- 이전
- 고동연 교수 / 탄소, 이제는 '제거'가 시급한 시대다
- 2023-11-27
- 다음
- 이현주 교수 / She did it '촉매 연구로 탄소중립을 이끌다'
- 2023-11-20

